Revisamos el seminario web y el panel de discusión de Pruebas genómicas con Dara Aisner, MD PhD, George Green, PhD y Greg Tsongalis, PhD. Con tanta información rica, estaremos publicando dos blogs. La Parte 1 cubrirá temas importantes seleccionados discutidos por cada orador, y la Parte 2 revisará las preguntas y respuestas de la audiencia.
Recientemente, tuve el placer de participar en un seminario web copatrocinado por LGC SeraCare y GenomeWeb. El tema fue “Pruebas genómicas para respaldar nuevas terapias para el cáncer avanzado” .
Esta fue una oportunidad para todos nosotros de escuchar a tres líderes de opinión (Dr. Dara Aisner, Dr. George Green y Dr. Greg Tsongalis) que han contribuido a la adopción constante de NGS pero que también conocen los muchos desafíos técnicos y clínicos. todavía nos enfrentamos para garantizar que todos los pacientes con cáncer tengan el diagnóstico correcto y el mejor tratamiento disponible. El formato del seminario web permitió presentaciones de cada panelista seguidas de debates y preguntas y respuestas de la audiencia.
Dra. Dara Aisner, MD PhD. fue el primer presentador. El Dr. Aisner es un patólogo anatómico y genético molecular e informático clínico certificado por la junta. Es profesora asociada de patología en la Facultad de Medicina de la Universidad de Colorado en Denver, Colorado, y miembro del Centro Integral del Cáncer de la Universidad de Colorado. Ha sido miembro de numerosos comités de pautas y patología oncológica, incluidos CAP/AMP/IASLC y National Comprehensive Cancer Network NCCN.
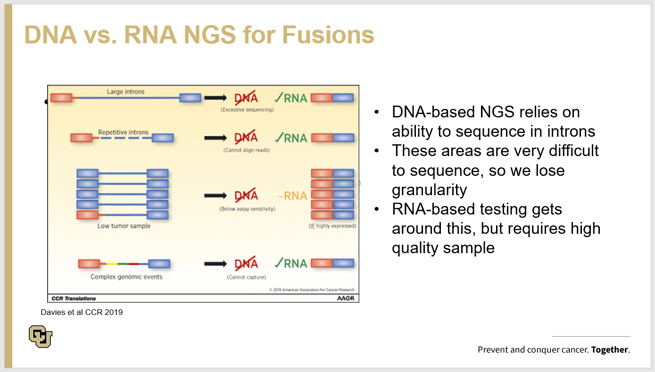
Durante su presentación, la Dra. Aisner usó un estudio de caso de cáncer de pulmón para mostrar cuán importante es garantizar que el tumor primario se use para evaluar exhaustivamente las fusiones de ARN si la mutación conductora no se identifica mediante el análisis de ADN NGS. El estudio de caso mostró cómo un panel NGS de 30 genes no pudo identificar la mutación conductora y el paciente fue seleccionado para inmunoterapia basada en PD-L1 IHC pero, lamentablemente, el cáncer del paciente progresó después de 6 meses. Después de la progresión, un flujo de trabajo de ARN de NGS identificó una fusión de ROS1 y se inició un fármaco inhibidor de ROS1 y las imágenes posteriores al tratamiento anti-ROS1 mostraron claramente la eliminación del tumor. El gráfico que se presenta a continuación muestra las deficiencias técnicas para detectar fusiones patogénicas solo con ADN y por qué se prefieren los flujos de trabajo de ARN. El Dr. Aisner también señaló que en ambos flujos de trabajo, no hay forma de evitar un problema con una muestra limitada o un rendimiento insuficiente del ácido nucleico extraído. Para obtener más información sobre su trabajo, consulte esta referencia que detalla las ventajas de detectar la fusión de ARN con una cantidad finita de muestra de tumor. (1)
La siguiente presentación estuvo a cargo del Dr. George A. Green. El Dr. Green es Director Ejecutivo y Jefe de Ciencias de Farmacodiagnóstico – Medicina Traslacional en Bristol Myers Squibb. Ha jugado un papel clave en el establecimiento de farmacodiagnósticos como una capacidad central dentro de BMS. El Dr. Green dirigió programas de desarrollo en oncología, neurociencia, cardiovascular y virología, así como programas de desarrollo de diagnóstico temprano en múltiples áreas terapéuticas y plataformas tecnológicas.
En el seminario web, el Dr. Green destacó la importancia de la monitorización de la enfermedad residual mínima (MRD) basada en biopsia líquida (ctDNA) para avanzar en la selección de pacientes en ensayos clínicos y para decisiones de tratamiento tempranas. El Dr. Green presentó datos de seguimiento de ctDNA de estudios clínicos publicados y fue bastante sorprendente ver que si el ADN del tumor es detectable en la sangre después del tratamiento inicial del cáncer, es altamente indicativo de la progresión de la enfermedad.
El Dr. Green también enumeró 12 ensayos clínicos en curso patrocinados por múltiples organizaciones donde los ensayos de MRD de ctDNA se encuentran en ensayos prospectivos en curso que evalúan los niveles de ctDNA con la respuesta al tratamiento en enfermedades en etapa temprana. Uno de los ensayos de fase III de CRC enumerados fue MGH 18-357 “ Identificación temprana y tratamiento de la enfermedad metastásica oculta en el cáncer de colon en estadio III ”. Tenía curiosidad por saber más sobre el ensayo y la indicación de ctDNA. Aquí está la descripción del ensayo. https://clinicaltrials.gov/ct2/show/NCT03803553 y la siguiente declaración se cita directamente del estudio.
“Después del diagnóstico y la extirpación quirúrgica de los tumores, las personas con cáncer colorrectal metastásico comúnmente reciben lo que se denomina tratamiento de quimioterapia adyuvante, que comúnmente utiliza planes de tratamiento llamados FOLFOX, CAPOX o terapia con 5-fluorouracilo.
Si no se elimina todo el cáncer, los investigadores pueden detectar un tumor en la sangre llamado ADN tumoral circulante (ADNct). Este es material genético exclusivo del cáncer colorrectal metastásico que puede estar presente en el torrente sanguíneo y puede identificarse mediante un análisis de sangre ctDNA. Si el ctDNA está presente en el torrente sanguíneo, comúnmente se denomina enfermedad micrometastásica (es decir, enfermedad que no se puede detectar mediante tomografías computarizadas, pero que puede estar presente en la sangre). Los investigadores del cáncer creen que el ctDNA en el torrente sanguíneo puede ser un indicador de que el cáncer tiene más probabilidades de reaparecer”.
….y del componente de investigación del ensayo.
- “Los investigadores están determinando si hay diferencias en la recurrencia del cáncer en los participantes positivos para ctDNA tratados con terapia adicional versus los que están en vigilancia activa.
- determinar si hay diferencias en la salud de los participantes positivos para ctDNA tratados con terapia adicional versus sometidos a vigilancia activa.
- examinar si los pacientes que se someten a una terapia adicional experimentan cambios en los niveles de ctDNA”.
Con respecto a los biomarcadores genotípicos, la cuantificación y los límites de detección, el protocolo establece que se genotiparon todos los tumores y que los biomarcadores genotípicos de inclusión primaria son deficiencia de reparación no coincidente documentada por IHC o expresión de proteínas o PCR (MSI-H), ctDNA en sangre positivo y ctDNA en sangre Se siguen las mutaciones BRAF V600E. Los objetivos de la investigación incluyen la medición de tendencias por debajo de un valor de umbral detectable. Los investigadores seleccionaron el ensayo LUNAR ctDNA de Guardant Health. Se ha informado que la sensibilidad analítica del 95 % del LoD de este ensayo es del 0,3 % de VAF para una entrada de ADN de 10 ng y <0,1 % de VAF para 30 ng.(2)
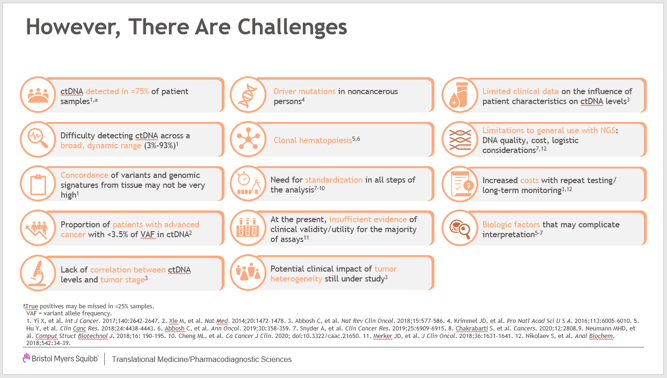
El Dr. Green cerró su presentación proporcionando un cuadro que muestra muchos desafíos que tenemos al aplicar las pruebas de MRD. Estos desafíos, como señaló, deben abordarse para seguir avanzando en la MRD para la atención del paciente.
El tercer panelista experto fue el Dr. Greg Tsongalis. El Dr. Tsongalis es el Director del Laboratorio de Genómica Clínica y Tecnología Avanzada (CGAT) en el Centro Médico Dartmouth-Hitchcock y el Centro de Cáncer Norris Cotton (NCCC). También es profesor y vicepresidente de investigación en el Departamento de Patología y Medicina de Laboratorio de la Facultad de Medicina Audrey and Theodor Geisel de Dartmouth. El Dr. Tsongalis fue presidente de la Asociación de Patología Molecular (AMP).
El Dr. Tsongalis y su equipo tienen muchos años de experiencia y una excelente reputación en la incorporación de nuevas tecnologías moleculares al laboratorio clínico a través de una validación rigurosa, sin comprometer nunca la calidad. En consecuencia, estaba bastante interesado en su experiencia evaluando y aplicando paneles de genes NGS a la atención de pacientes. Greg informó que su laboratorio usó un panel de puntos críticos de cáncer de 50 genes durante 6 años y debido a que se validaron clínicamente nuevos genes impulsores y se aprobaron nuevos tratamientos, validaron y adoptaron un panel de genes ILMN TST170 en 2020. Después de solo 6 semanas implementando el panel de 170 genes para respaldar las pruebas clínicas de los pacientes, los resultados fueron bastante impresionantes. El laboratorio secuenció un 25 % más de tumores de los que habría secuenciado el panel de 50 genes, detectando 12 fusiones adicionales, 3 mutaciones de omisión MET ex14,
De manera similar al cargo del Dr. Green para todos los que estamos involucrados en la genómica clínica, el Dr. Tsongalis enumeró los muchos desafíos que enfrentan los laboratorios moleculares en la actualidad. Vea el gráfico a continuación.

Además de los desafíos enumerados anteriormente, el Dr. Tsongalis ha respondido a la solicitud constante de las juntas de tumores para ayudar a reducir el tiempo de respuesta (TAT) que informa los resultados genotípicos para acortar el período desde el diagnóstico hasta el tratamiento. Para aquellos que estén interesados en obtener más información sobre este tema, consulte Tsongalis et al. (3) Aquí evaluaron las mutaciones de NRAS, KRAS y BRAF dirigidas a CRC. El estudio comparó el enfoque estándar de atención de un laboratorio (secuenciación de Sanger o NGS o qPCR) con la plataforma totalmente automatizada Biocartis Idylla para 780 casos para determinar el informe de resultados de la prueba TAT en comparación con los ensayos estándar. La PCR tuvo un TAT de prueba promedio de 5,6 días, la PCR de envío fue de 22,5 días, la secuenciación de Sanger interna fue de 14,7 días, la de Sanger de envío fue de 17,8 días, la NGS interna de 12,5 días y la NGS de envío fue de 20,0 días. Las pruebas estándar tuvieron un tiempo de respuesta promedio de 11 días. Idylla promedió TAT de 4,9 días.
Concluí la parte de presentación del seminario web revisando los materiales y estándares de referencia de NGS que desarrollamos y fabricamos en LGC Seracare para oncología clínica. Estos incluyen perfiles de tumores, biopsia líquida e inmuno-oncología. También aproveché esta oportunidad para presentar varios productos nuevos de Seraseq Oncology lanzados recientemente:
Inmuno-Oncología:
- Seraseq Blood TMB, puntuaciones de 7, 13, 20, 26 al 2 %, 0,5 % y 0 % para el desarrollo, la validación y el control de calidad del ensayo
DMR:
- Seraseq ctDNA MRD Panel Mix, consta de una línea celular compatible con tumor normal más 25 construcciones de ADN de variantes clínicamente relevantes en 15 genes, fragmentadas, dimensionadas y mezcladas en fracciones tumorales de 0,5 %, 0,05 %, 0,005 % y 0 % para ctDNA NGS MRD desarrollo de ensayos, validación, QC y PT/EQA
Perfiles de tumores:
- Seraseq Lymphoma Formatos FFPE y gDNA, SNV y del en BCL2, BRAF, DNMT3A, EZH2, IDH2, MYD88, CXCR4, NOTCH1,2, RHOA, SF3B1, STAT3, STAT5B, TP53, 5-15 % VAF
- Seraseq Whole Transcriptome RNA Seq mix, multiplexado a 22 detección de fusión de ARN mediante ensayos NGS de transcriptoma RNA Seq.
Infórmate sobre otros temas con nuestro blog. Visita nuestras redes sociales para más información


