Los flujos de trabajo del análisis de perfiles tumorales comienzan a partir de una muestra de biopsia de tejido que se fija con formalina e incrustada en parafina (FFPE), un proceso que introduce varios tipos de daño en los ácidos nucleicos de la muestra de tejido. Faltan materiales de referencia que imiten fielmente el perfil de daño de las muestras de FFPE de pacientes. En el ADN extraído de tejido FFPE se puede encontrar depurinación, despirimidinación, desaminación, oxidación, mellas y roturas de doble hebra, a pesar del uso de kits de extracción que intentan reparar parte de este daño. Hemos desarrollado un material de referencia biosintético multiplexado y dañado con formalina, Seraseq® Compromised FFPE Tumor DNA, así como un material complementario de tipo salvaje (WT), Seraseq Compromised FFPE WT, para imitar el daño encontrado en las muestras de pacientes que se pueden utilizar para evaluar todo el flujo de trabajo de elaboración de perfiles de tumores. Comparamos el rendimiento de estos materiales de referencia en ensayos posteriores con el del material FFPE con daño mínimo en el ADN, como el producto Seraseq FFPE WT (ADN / ARN) RM.
Fabricación de material de referencia
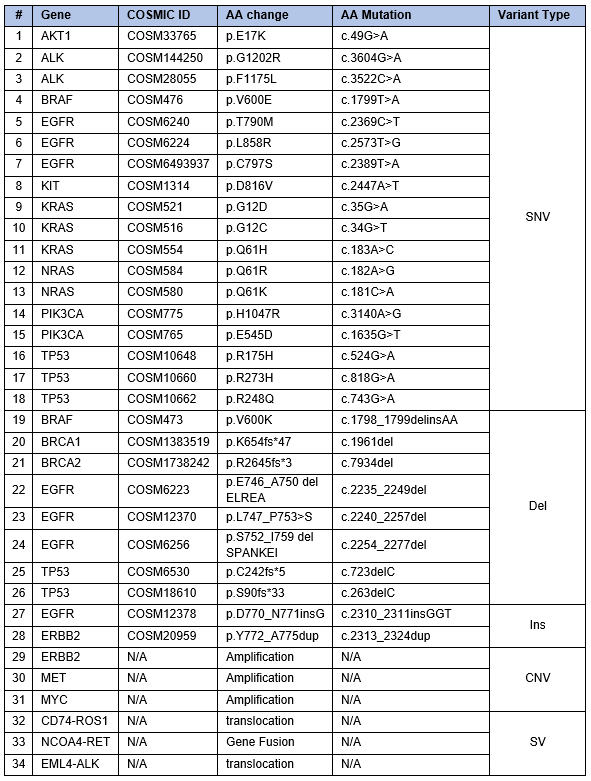
Los materiales de ADN tumoral FFPE comprometido de Seraseq se fabricaron modificando la línea celular de referencia GM24385 con ADN biosintético que cubre 34 variantes, incorporando todos los tipos de variantes: SNV, INDEL, CNV y SV (Tabla 1). Las células manipuladas se diluyeron con células WT para lograr las frecuencias alélicas deseadas y los números de copias determinados por PCR digital. Esta mezcla de células, así como las células WT solas, se procesaron con un protocolo FFPE patentado que introduce daño moderado o con un protocolo FFPE de fijación ligera.
Tabla 1. Variantes encontradas en el material de referencia de ADN tumoral FFPE comprometido de Seraseq.
Comparación de rendimiento y calidad
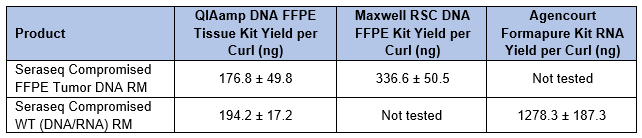
Se utilizaron varios kits de extracción de FFPE para aislar ADN y / o ARN de secciones de 10 µm de cada producto (Tabla 2). Los rendimientos fueron similares y suficientes para los procesos posteriores, independientemente del método utilizado para la fijación. Tabla 2. Rendimientos de ADN y ARN de materiales de referencia FFPE comprometidos de Seraseq utilizando kits de extracción disponibles comercialmente.
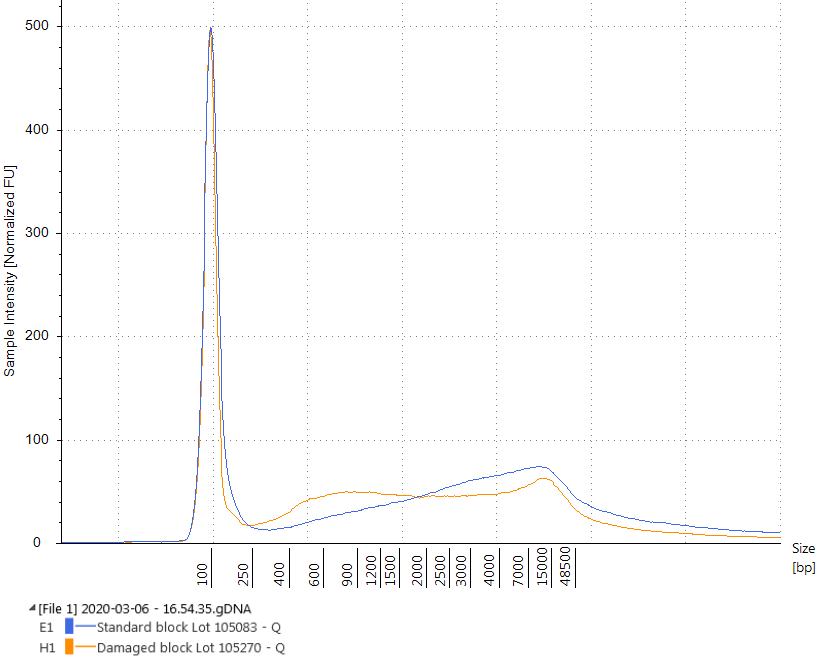
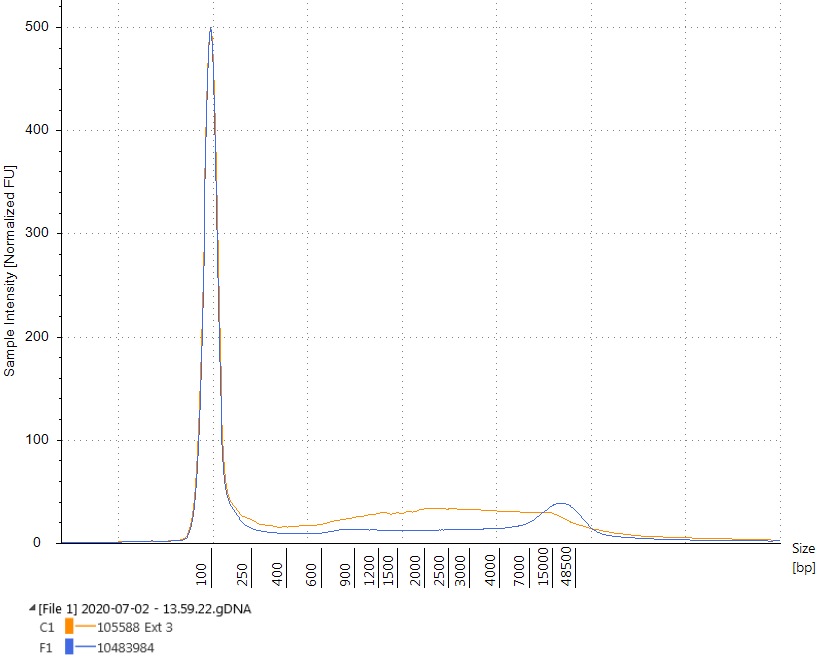
La calidad del ADN de los ácidos nucleicos extraídos se evaluó mediante Agilent TapeStation. La calidad del ADN extraído con el QIAamp DNA FFPE Tissue Kit de cada tipo de bloque se evaluó utilizando el TapeStation Genomic DNA Kit, que calcula el número de integridad del ADN para cada muestra en un rango de 1 a 10, donde una puntuación de 10 indica ADNg intacto. El proceso de fijación moderado utilizado para los materiales de ADN tumoral de FFPE comprometido da como resultado valores de DIN que son un 28,9% más bajos (valor de DIN medio de 3,98 ± 0,27 frente a 5,60 ± 0,14) que los valores de DIN del ADN extraído de los rizos de FFPE realizados mediante el proceso de fijación por luz. (Figura 1). De manera similar, los materiales FFPE WT comprometidos tenían valores DIN más bajos que sus contrapartes no comprometidas (valor DIN promedio de 3,95 ± 0,17 frente a 5,87 ± 0,17, una reducción del 32,7%) como se ve en la Figura 2.
Estos datos indican que modificamos con éxito la cantidad de daño en el material. El valor DIN promedio de nuestro material de fijación moderado está cerca del ADN promedio extraído de una muestra de paciente usando el mismo kit de extracción y da como resultado un valor DIN de 3.31.
La calidad del ARN fue baja para ambos métodos de fabricación de FFPE, con puntuaciones de RIN medidas de ~ 1,0 para el material extraído de los rizos Seraseq Compromised WT y Seraseq FFPE WT con el kit Agencourt Formapure, medido con el kit Agilent Bioanalyzer RNA 6000 Nano. Estos valores son similares a los valores de RIN observados para el ARN extraído del FFPE del paciente (promedio 1,7 ± 0,5) utilizando el mismo kit2.
Análisis de variantes
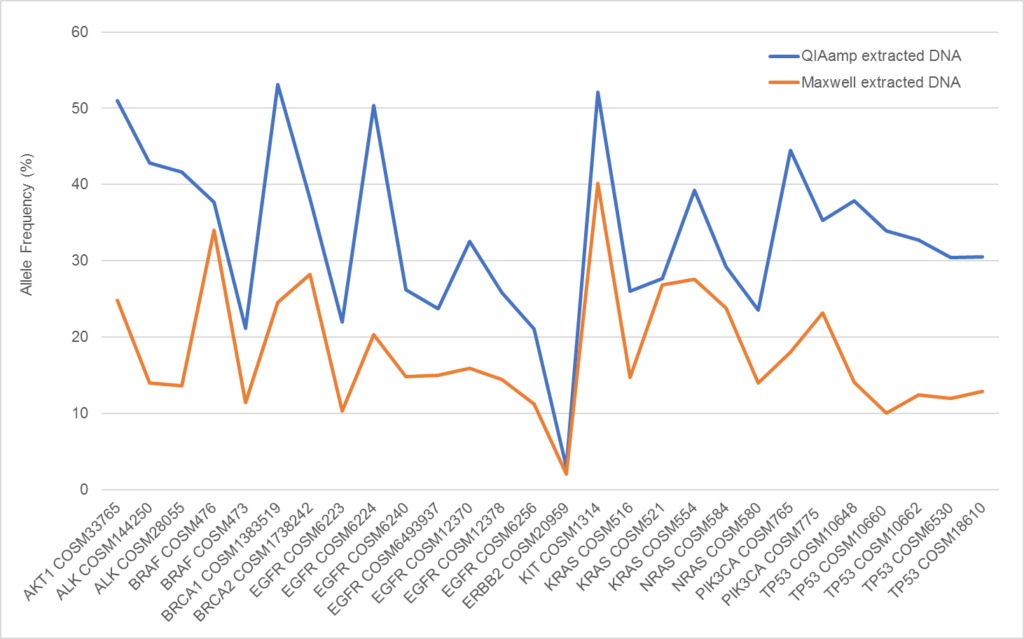
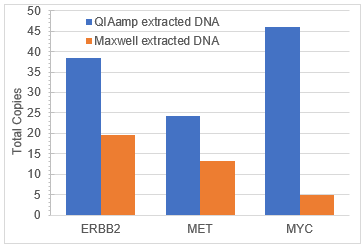
Cuando se realizó el análisis de variantes con el ensayo Illumina TSO500, se observó una VAFS diferencial entre el ADN extraído del mismo bloque de ADN tumoral FFPE comprometido con Seraseq con diferentes kits de extracción de FFPE (Figura 3). Es probable que esta variación se deba tanto a la variabilidad en la eficiencia de extracción del ADN genómico como a las variantes biosintéticas agregadas, así como a las diferencias en la actividad bioquímica y al sesgo en la reversión del daño del formaldehído durante el aislamiento del ADN con los kits respectivos. La variación en la VAF aparente debido al método de extracción es un fenómeno conocido1. Todas las variantes se detectaron entre el 2% y el 55%. De manera similar, los números de copias medidos para cada gen amplificado fueron variables (Figura 4), pero bien respaldados en cada muestra. Se observaron resultados similares cuando el ADN extraído se analizó mediante PCR digital (datos no mostrados).
Como era de esperar, la secuenciación de ADN y ARN utilizando el ensayo ArcherDx VariantPlex Solid Tumor Assay y el ArcherDx FusionPlex Solid Tumor Assay, respectivamente, no reveló variantes patógenas en el material de referencia Seraseq Compromised FFPE WT ni en el material de referencia Seraseq FFPE WT cuando las muestras se secuenciaron a una profundidad de ~ 1 millón de lecturas por muestra. Si bien las longitudes promedio de los fragmentos fueron más cortas para el material comprometido que para el material de fijación de luz (178.0 bp vs 196.8 bp para ADN y 146.9 bp vs 159.5 bp para ARN), ambos materiales se desempeñaron bien en los ensayos, con concentraciones de biblioteca suficientes (> 6 nM para ADN,> 26 nM para ARN), lecturas alineadas y mapeadas altas (> 99% para ADN y ARN) y lecturas adecuadas en el objetivo (> 98% para ADN y> 94% para ARN).
En general, ambos métodos FFPE dan como resultado materiales de referencia que son compatibles con los ensayos de PCR y NGS (basados en amplicones y captura híbrida). Con productos comprometidos, el rendimiento puede verse afectado por el método de extracción FFPE implementado para recuperar ADN amplificable para el análisis posterior. Por tanto, el material de referencia de ADN tumoral comprometido de Seraseq servirá bien como un producto cualitativo para evaluar la capacidad de un ensayo para detectar las variantes incluidas.
El método de fijación moderada imita más de cerca las biopsias de tejido archivadas de pacientes con cáncer a la vez que mantiene la capacidad de multiplexación y el rendimiento posterior, lo que hace que estos productos Seraseq sean aplicables en el desarrollo de ensayos de NGS, la optimización de la tubería de bioinformática y como referencias de procesos completos en ensayos clínicos de NGS utilizados para pruebas de pacientes.
Referencias:
- Bonnet E, Moutet M-L, Baulard C, et al. Comparación del rendimiento de tres kits de extracción de ADN en datos de exoma completo humano de muestras normales y tumorales incluidas en parafina fijadas con formalina. Más uno. 2018; 13 (4); e0195471.
- Kresse SH, Namløs HM, Lorenz S, Berner J-M, Myklebost O, Bjerkehagen B y Meza-Zepeda LA. Evaluación de métodos comerciales de extracción de ADN y ARN para muestras de FFPE de secuenciación de alto rendimiento. Más uno. 2018; 13 (5); e0197456.
Temas: NGS


